
2021年10月6日,省部共建非人灵长类生物医学国家重点实验室/昆明理工大学灵长类转化医学研究院季维智院士团队、牛昱宇课题组与哈佛医学院张毅课题组合作在 Developmental Cell杂志上发表了题为“Analysis ofdevelopmental imprinting dynamics in primates using SNP-free methods identifyimprinting defects in cloned placenta”的研究工作。该工作成功揭示了灵长类早期胚胎、成体细胞以及胎盘组织间基因印记的差异,并由此发现了克隆猴胎盘中的基因印记存在严重缺失。为远交系动物基因印记研究提供了高效精准的方法,也为提高灵长类动物的克隆效率带来了新的思路。
基因印记是双亲来源的染色体中单个等位基因表达的现象,在多种生物学过程中发挥着重要作用[1]。但目前,鉴定人类等远交系动物(outbred animals)的印记区域需要使用基于单核苷酸多态性(Single nucleotide polymorphisms, SNP)的分析方法并整合数百个不同个体的DNA甲基化组数据[2]。这类方法成本高昂且材料不易获得,这成为了在远交系动物中研究基因印记的一大障碍。
为了能更好的了解灵长类动物的基因印记调控机制,作者首先对食蟹猴16细胞时期的孤雄与孤雌胚胎做了转录组测序并鉴定出371个父源高表达基因(paternal-biased expressed genes, PEGs)。并通过DNA甲基化测序和组蛋白甲基化(H3K27me3)数据分析发现在这371个PEGs中,144个与早期胚胎母源高甲基化区域(early-embryonic maternal-allele-methylateddifferentially methylated regions,emDMRs)相关而仅有7个与母源高H3K27me3区域相关。这表明,与啮齿类动物不同,灵长类早期胚胎中PEGs的表达主要受到DNA甲基化而非H3K27me3的调控。
为确定早期胚胎中与PEGs相关的emDMRs是否会被保留至成体组织中,以及确定胚外组织来源的胎盘中的印记区域与其他体细胞组织的印记区域是否存在差别,作者分别开发了不依赖SNP鉴定印记区域的方法—TARSII (Tissue-associated, reads-based, SNP-freemethod for identifying imprint-DMRs)以及用于预测单一组织中印记区域的方法—CARSII(CpG-island-associated, reads-based, SNP-freemethod for identifying imprint-DMRs)。并最终发现大部分早期胚胎时期的emDMRs并不会维持到成体组织中也不会维持到胚外来源的胎盘组织中。此外基于CARSII分析发现,人、食蟹猴与猕猴胎盘中的母源印记区域要远多于成体组织中的数量(约为3倍),而这种现象在小鼠组织中没有被观察到,表明这是一种灵长类特有的胎盘印记模式。
考虑到核移植动物的供体细胞也是体细胞,是否核移植胎盘中的印记模式能够与野生型胎盘的保持一致?作者进一步预测了核移植胎(SCNT)胎盘印记区域并与野生型胎盘进行比较,发现并证实了核移植胚胎的胎盘丢失了大量该有的基因印记,且这些印记区域对应的基因的表达也存在异常(图1)。有趣的是,这些异常的基因中有的被认为对胚胎和胎盘的发育具有重要的意义,如DNMT1和HAND2。因此克隆猴胎盘中大量印记的丢失很有可能是造成克隆猴胚胎发育率极低的重要原因之一。
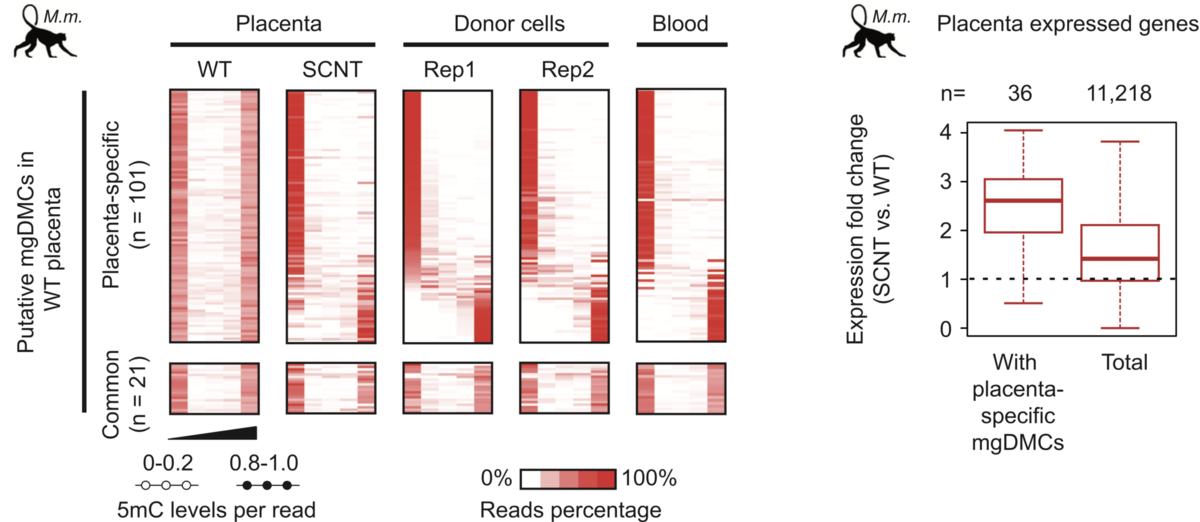
图1. 野生型、核移植胎盘和成体组织中胎盘特异母源印记DMRs DNA甲基化修饰水平(左)及对应基因的表达情况(右)
综上所述,本研究探索并揭示了灵长类动物从早期胚胎至出生后的基因印记动态图谱。并证实了体细胞组织并不能维持早期胚胎的基因印记模式,且这种体细胞印记模式并不能亦或者很难通过核移植重编程被逆转,从而导致了核移植胎盘的基因印记缺陷最终影响了核移植胎盘和胚胎的发育(图2)。本项工作为灵长类动物核移植研究提供了重要的线索,也为提高灵长类动物的克隆效率带来了新的思路。
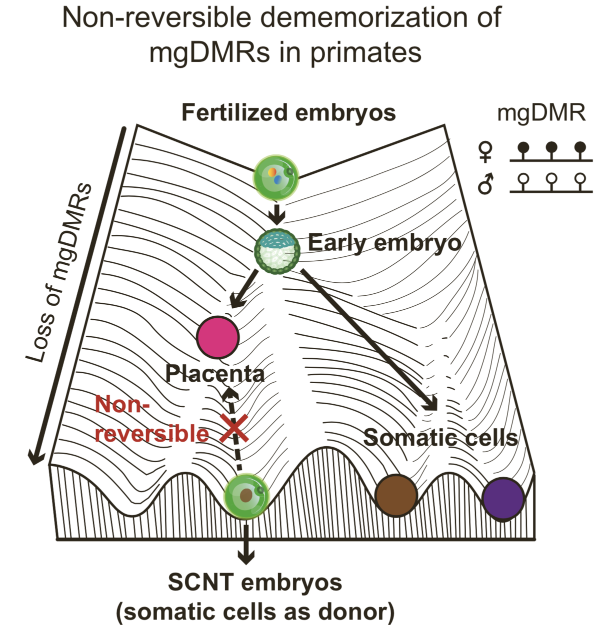
图2. “mgDMR不可逆擦除”模型
参考文献
1. Tucci,V., et al., Genomic Imprinting andPhysiological Processes in Mammals. Cell, 2019. 176(5): p. 952-965.
2. Zink, F., et al., Insights into imprinting from parent-of-originphased methylomes and transcriptomes. Nat Genet, 2018. 50(11): p. 1542-1552.
